Ein „antikes“ T-Shirt aus den Tiefen des Kleiderschranks erinnert an „Epigenetik 2003“.

Das war eine der ersten größeren Epigenetik-Konferenzen, die im September 2003 als Jahrestagung der Gesellschaft für Genetik stattfand. Nur einen Monat vorher gab es, zum selben Thema und teilweise mit denselben Sprechern in Holderness, USA eine Gordon Research Conference.

Was versteht man unter Epigenetik?
Vor 20 Jahren waren die grundsätzlichen Bereiche der Epigenetik festgelegt:
Veränderungen in der Ausprägung der genetischen Information, ohne dass die Information, d.h. die DNA Sequenz selbst verändert wurde.
Dazu gehören
- Kleine Veränderungen der DNA, wie das Anhängen einer Methylgruppe an die Base Cytosin.
- Kleine Veränderungen an den Histonproteinen, dem Verpackungsmaterial der DNA.
- Die Entdeckung „kleiner regulatorischer RNAs“ die lange Zeit für nutzlose Bruchstücke und „Abfall“ gehalten wurden.
Diese drei Faktoren verändern nicht die genetische Information der DNA, sie verändern die „Lesbarkeit“, d.h. wann und wie stark ein Gen abgelesen und in Protein übersetzt wird. Durch epigenetische Kontrolle können Gene in verschiedenen Geweben hoch- oder runtergefahren oder auch ganz ausgeschaltet werden.
DNA Methylierung
Eine methylierte Cytosin-Base wird bei der Übersetzung in Protein genauso gelesen, wie eine normale Cytosin-Base, aber sie erschwert meistens den Zugriff der Transkriptionsmaschinerie. Das bedeutet, der Text eines Gens ist fehlerfrei vorhanden aber es liegt ein mehr oder weniger dunkler Balken darüber, sodass er nur schwer oder gar nicht gelesen werden kann.
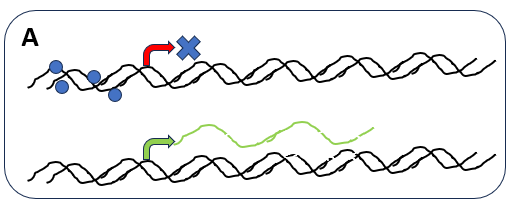
Oben: der DNA-Strang ist vor einem Gen methyliert (blaue Kreise). Am Start der Transkription (roter Pfeil) kann das Enzym zum Ablesen des Gens schlecht oder nicht ansetzen. Das Gen ist stumm geschaltet.
(Abb.: BioWissKomm)
Unten: der DNA-Strang ist nicht methyliert und vom grünen Pfleil aus kann das Gen als RNA abgelesen werden (grüne Linie).
Histone – die Verpackung der DNA
Die Familie der Histonproteine (Histone) verpackt die DNA zu einem Komplex, dem Chromatin und hilft, den sehr langen DNA Faden in kompakter Form geordnet im Zellkern unterzubringen. Dabei bilden i.d.R. vier verschiedene Histone (H2A, H2B, H3 und H4) ein Nukleosom, eine Art Kugel, um die die DNA aufgewickelt ist (in Abb. B als blauer Kreis vereinfacht dargestellt. Als Verbindungsstück zwischen den Nukleosomen kommt noch das Linker-Histon H1 hinzu. Diese Verpackung muss jedoch gelockert werden, um einen DNA Abschnitt (z.B. ein Gen) abzulesen und eine entsprechende mRNA herzustellen. Kleine chemische Gruppen können an verschiedene Stellen der Histonproteine angehängt werden und erleichtern oder erschweren das Ablesen der DNA in RNA. Diese Anhängsel oder Modifikationen sind sehr komplex. Es gibt viele verschiedene davon (z.B. Phospahtgruppen, Acetylgruppen, Methylgruppen), die an verschiedene Bausteine (Aminosäuren) der Histonproteine angehängt werden können. Dadurch entsteht der „Histon-Code“ der in verschiedenen Geweben zu verschiedenen Zeiten und unter verschiedenen Bedingungen einen DNA Abschnitt mehr oder weniger „auflockert“ und zugänglich für das Ablesen macht. In manchen Fällen müssen Nukleosomen verschoben, gegen Varianten (s.u.) ausgetauscht oder ganz abgelöst werden. Dafür gibt es spezielle „Chromatin-Modifier“, Enzyme, die auf der DNA die Nukleosomen rearrangieren können.
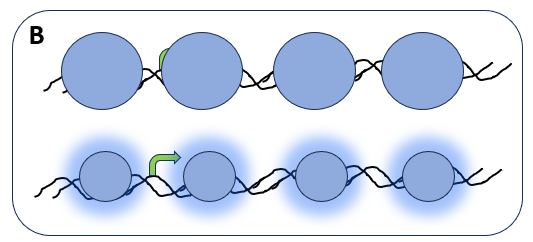
DNA und Histon-Proteine bilden zusammen das Chromatin, einen Komplex, der die DNA verpackt und schützt. Durch kleine chemische „Anhängsel“ wird das Chromatin kompakt und verdeckt die Startpunkte für das Ablesen einzelner Gene (oben). Durch andere chemische „Anhängsel“ wird das Chromatin aufgelockert. Die Startpunkte für das Ablesen von Genen werden zugänglich (unten).
(Abb.: BioWissKomm)
Zusätzlich gibt es einige „Spezialhistone“ oder Histonvarianten, die an besonderen Stellen eine besondere Form von Chromatin bilden. So ersetzt „Macro-H2A“ das gewöhnliche H2A teilweise am inaktivierten X-Chromosom weiblicher Säuger. (Säugerweibchen haben zwar zwei X-Chromosomen, eins davon ist aber weitgehend inaktiv und als „Barr-Körper“ so fest verpackt, dass es kaum abgelesen werden kann.) Das Centromer ist an jedem Chromosomen der Ansatzpunkt für die Spindelfaser. Sie verteilen bei der Zellteilung die Chromosomen gleichmäßig auf die Tochterzellen. Auch hier gibt es spezielle Nukleosomen, die statt des üblichen Histons H3 die Variante CENP-A enthalten.
Kleine RNAs – wichtige Winzlinge
Kleine regulatorische RNAs wurden erst 1993 im Fadenwurm C. elegans entdeckt und später als Mikro-RNAs (miRNAs) bezeichnet. Sie werden nicht von „richtigen“ Genen abgelesen, die für mRNAs codieren, sondern von anderen DNA Abschnitten, denen man bis dahin keine Aufmerksamkeit geschenkt hatte. Mikro-RNAs können an mRNAs binden und die Translation (die Übersetzung in Proteine) blockieren. Nachdem sie einmal bekannt waren, fand man tausende von miRNAs in nahezu allen Organismen und erkannte ihre wichtige Rolle bei der Genregulation. Hinzu kam weiter: siRNAs, piRNAs und andere, die ähnliche Funktionen haben.
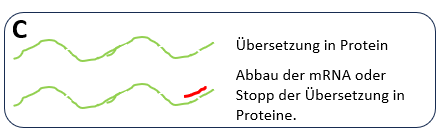
mRNAs (grün) sind vorübergehende Kopien von Genen, die in Protein übersetzt (translatiert) werden sollen. Danach werden sie abgebaut. Das kann durch kleine siRNAs bewikt werden (nicht gezeigt). Manche mRNAs werden länger auf Vorrat aufgehoben aber ihre Translation wird durch miRNAs (unten, rot) verhindert. Erst wenn die Proteine gebraucht werden, werden die mRNAs wieder freigegeben.
(Abb.: BioWissKomm)
Du bist was du isst?
Besonders die DNA Methylierung hat zu aufsehenerregenden Entdeckungen geführt. Bei unserem Kongress 2003 berichtete Randy Jirtle über brandneue Ergebnisse mit speziellen, gentechnisch veränderten Mäusen in denen er durch Fütterung einer trächtigen Maus mit Folsäure ein konstruiertes Gen ausschalten konnte. Der Phänotyp, d.h. das Aussehen der Nachkommen änderte sich dramatisch: ohne Folsäure waren sie fettleibig und hatten ein helles Fell, mit Folsäure waren sie schlank und hatten das normale braune Fell.
Es stellte sich heraus, dass die Fütterung dazu führte, dass das Gen, welches zu Fettleibigkeit und hellem Fell führte, durch DNA-Methylierung abgeschaltet wurde. Dies war ein kompliziert konstruiertes Beispiel. Es führte aber zu der Folgerung, dass Ernährung durchaus einen Einfluss auf die Gene haben kann.

Die dicke und die dünne Maus unterscheiden sich nur durch die Ernährung der Mutter, sonst sind sie genetisch identisch. (Abb. © BioWissKomm by Midjourney).
„Du bist was du isst“ enthält gewiss einen Funken Wahrheit, aber ganz so einfach ist es dann doch nicht! Ernährung und andere Umwelteinflüsse wirken mit Sicherheit auf das Epigenom, aber ein Nahrungsergänzungsmittel aus dem Supermarkt wird uns sicherlich nicht so schnell schlank, agil und gesund machen!
Anwendungen
Es gibt viele Projekte, das Epigenom sorgfältig zu untersuchen und Ansatzpunkte zur Behandlung von Erkrankungen zu finden.
Erfolge gibt es bereits mit dem Krebsmedikament Azacitin, das die DNA-Methylierung reduziert. https://www.newsletter-epigenetik.de/zeitenwende-in-der-leukaemie-therapie/. Ein Werkzeug, das aus CRISPR-Cas entwickelt wurde, erlaubt heute die Methylierung von DNA an beliebigen Stellen des Genoms und so einen gezielten Eingriff in das Epigenom. Wie das funktioniert, zeigen wir in einem leicht verständlichen Video (englisch).
Kleine RNAs waren 2003 noch sehr neu. Inzwischen hat man tausende davon gefunden und ihre verschiedenen Funktionsweisen ziemlich gut verstanden.
2018 wurde in den USA das erste Medikament zugelassen, das auf einer siRNA (small interfering RNA) beruht.
Auch in der Pflanzenzucht spielen kleine RNAs eine große Rolle und finden bereits Anwendung: sie können Gene ausschalten die für den Ertrag unvorteilhaft sind oder den Nährwert eines landwirtschaftlichen Produkt verringern. Geradezu genial sind erste Versuche, kleine RNAs gegen Schadinsekten einzusetzen. Damit könnte der Verbrauch an Pestiziden drastisch reduziert werden.
Diagnostik
DNA-Methylierung, Histon-Modifikation und kleine RNAs sind auch zu wichtigen diagnostischen Werkzeugen geworden. Man kennt Muster, die als Bio-Marker für verschiedene Krankheiten dienen. Mit modernen molekularbiologischen Methoden können sie relativ einfach in Gewebeproben analysiert werden und erlauben z.B. eine deutlich frühere Erkennung von Krebs – und damit eine deutlich bessere Prognose bei der Therapie.
Eine von vielen Methode, wie man z.B. die Gesamtheit kleiner RNAs in Zellen oder einem Gewebe erfassen kann, haben wir hier beschrieben.
Die Zukunft
Was 2003 noch hauptsächlich Grundlagenforschung war, ist heute ein innovatives Feld in der praktischen Anwendung auf vielen Gebieten. Dazu gehört auch die immer detailliertere Analyse des Epigenoms und wie es durch Umwelteinflüsse verändert werden kann. Biochemische und molekularbiologische Methoden werden entwickelt, um gezielt epigenetische Veränderungen zu erreichen, um z.B. Krebs zu heilen oder Pflanzen an ein sich ändernde Umwelt anzupassen. Aber auch in der Grundlagenforschung bringt die Epigenetik immer wieder Überraschungen: so fand man, dass sich Königin und Arbeiterinnen in einem Bienenvolk nur epigenetisch unterscheiden und dass dieser Unterschied ausschließlich durch das verschiedenen Futter bewirkt wird.
Hier gilt tatsächlich „du bist was du isst“ – aber ob sich das so auf den Menschen übertragen lässt, ist sehr fraglich!

Königin durch bessere Ernährung? Sicherlich gilt auch beim Menschen, dass Arme öfter an Hunger und Mangelernährung leiden als Reiche. Das ist aber eher auf direkte Effekte durch die Nahrung zurückzuführen. Die Bienen hat man jedoch gefunden, dass die Ernährung einen Einfluss auf das Epigenom hat. (Abb. © BioWissKomm by Midjourney).
Autor:
Wolfgang Nellen, BioWissKomm

