Nein, ganz so einfach ist das nicht! Aber hinter der Schlagzeile des epd (evangelischer Pressedienst) steckt mehr, als es die Kurzmitteilung in vielen Zeitungen vermuten lässt.
Aus einer Tiefsee Meta-Genom-Analyse konnte die Kristallstruktur einer Feruloyl-Esterase aus dem nicht-kultivierbaren Archaeon Candidatus Bathyarchaeota mit einer Auflösung von 1,71 Angström aufgeklärt werden.
Wie bitte? Was soll das denn heißen?
Seit Jahren sucht man nach Enzymen, die unseren Plastikmüll abbauen können und ist dabei auch fündig geworden: es gibt bisher etwa 80, die Polyethylene terephthalate (PET) mehr oder weniger gut abbauen können. Das neue Enzym ist für ein großtechnisches biologisches Recycling interessant, da es nicht nur das Polymer angreift, sondern äußerst aktiv zwei der beim Abbau anfallendem Zwischenstufen abbaut. Obwohl die Anwendung fest im Blick ist: zum größten Teil geht es noch um die Grundlagenforschung. Man fahndet nach Enzyme, die möglichst effizient Kunststoffe abbauen können und man versucht, sie nach bestem Wissen zu optimieren. Dazu braucht man möglichst viele davon, um ihre Struktur und Funktion zu verstehen. Das Wissenschaftskonsortium von den Universitäten Kiel, Hamburg und Düsseldorf hat dazu einen wesentlichen neuen Beitrag geleistet: sie haben ein sehr wirksames Enzym (PET 46) aus einem Tiefseeorganismus entdeckt, den man im Labor nicht wachsen lassen kann und den noch nie ein Mensch gesehen hat.
Wie soll das denn gehen?
Metagenome
Die Geschwindigkeit der DNA Sequenzierung und die Bioinformatik haben atemberaubende Fortschritte gemacht! Man kann heute aus einem Esslöffel Erde oder Schlamm, einem Liter Wasser oder sogar Luft ausreichende Mengen DNA isolieren und sequenzieren ohne zu wissen, was da alles drin ist – nach der Sequenzierung und der Datenanalyse weiß man etwas besser. Oder auch nicht, wenn man ungewöhnliche Proben, z.B. aus der Tiefsee analysiert.
Die Bioinformatik leistet dann Hilfestellung: durch Vergleiche mit riesigen Sequenzdatenbanken kann vielen unbekannten Gensequenzen aus unbekannten Organismen eine vermutliche Funktion zugeordnet werden. Die entsprechenden Proteine können anschließend in einem Labororganismus produziert werden und die Funktion (z.B. Abbau von PET) kann im Labor überprüft werden. Gleichzeitig wird die Struktur des Proteins untersucht: Wie sieht es aus? Wie arbeitet es? Was sind Ähnlichkeiten und Unterschiede zu bereits bekannten Proteinen?
Die „Dreckprobe“ in der PET46 entdeckt wurde stammt aus dem Guayamas Becken vor der Küste Süd-Kaliforniens. Dort wurden vom Tauchboot ALVIN in 2000m Tiefe Sedimente rund um heiße Tiefseequellen entnommen.
Die Arbeitsschritte für eine Metagenomanalyse sind in dem Flussdiagramm vereinfacht dargestellt
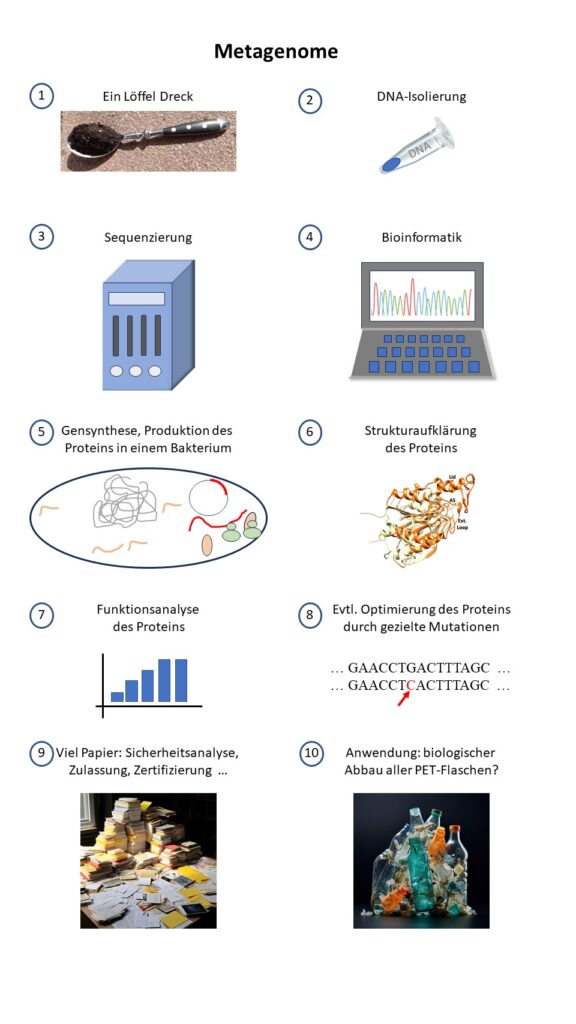
1. „Ein Löffel Dreck“ steht praktisch für alles. Organismen und ihre Überreste (und damit auch ihre DNA) findet man überall: in der Erde, im Schlamm, im Wasser, in der Luft und eben auch in Sedimenten aus der Tiefsee.
2. Mit sehr empfindlichen Methoden können auch kleinste Mengen an DNA aus solchen Proben gewonnen werden. Das Wichtigste dabei ist, sie nicht zu verunreinigen. Ohne super-saubere Chemikalien und Geräte würde man überwiegend menschliche DNA, Mikroben und andere DNA-Stücke aus der Laborumwelt finden.
3. Moderne Sequenziermaschinen können Millionen von DNA Stücken in kürzester Zeit parallel sequenzieren, d.h. die Abfolge der Nukleotide, des genetischen Alphabets, bestimmen. Diese Technik ist heute nahezu vollständig automatisiert.
4. Es war eine Herausforderung, diese Unmenge an Daten zu verarbeiten um etwas mit den ganzen Gs, As, Ts und Cs anfangen zu können. Auch dafür gibt es inzwischen mächtige Bioinformatikprogramme, die alle sequenzierten DNA-Stücke mit riesigen Datenbanken vergleichen. Einem großen Teil der Sequenzen können sie damit automatisch Gene und Funktionen zuordnen – alleine durch Ähnlichkeit mit bereits bekannten Sequenzen.
5. Hat man ein Gen von Interesse gefunden, kann es nach der Sequenz „geschrieben“, d.h. synthetisch hergestellt werden. Das synthetische DNA Stück wird in vielen Kopien in einen Labororganismus eingeschleust. Meist verwendet man das Bakterium E. coli, das „Arbeitspferd“ der Molekularbiologen. Dort wird das synthetische Gen, wie jedes andere auch, in RNA übersetzt und davon wird das entsprechende Protein hergestellt. Laborstämme von E. coli sind so gezüchtet, dass sie sehr große Mengen
des gewünschten Proteins produzieren. Sie liefern also genügend Material für weitere Experimente.
6. Dazu gehört die Strukturaufklärung, die mit technisch sehr anspruchsvollen Methoden wie der Röntgenstrukturanalyse, NMR (Kernspinresonanzspektroskopie) und anderen durchgeführt werden. Hinzu kommen bioinformatische Methoden und in jüngerer Zeit auch KI-Programme. Mit der Strukturaufklärung wird einmal die dreidimensionale Form eines Proteins bestimmt, d.h. wie die vom Gen codierte Aminosäurekette gefaltet wird und welche weiteren Verknüpfungen in der gefalteten Form vorliegen. Weiterhin kann man daraus auch Bewegungen des Proteins ableiten, z.B. wie ein Substrat erkannt, gebunden und in die richtige Position gebracht wird, um es enzymatisch zu verarbeiten.
7. In der Funktionsanalyse wird untersucht, welche Substrate z.B. ein Enzym verarbeiten kann (hier z.B. PET in verschiedenen Formen) und was die Endprodukte sind. Weiterhin werden die optimalen Arbeitsbedingungen bestimmt: bei welcher Temperatur, bei welchem pH-Wert, in
welcher chemischen Umgebung funktioniert das Enzym am besten?
8. Aufgrund der Struktur, langer Erfahrungen, Computervorhersagen und inzwischen auch KI können gut begründete Vermutungen angestellt werden, mit welchen Veränderungen ein Enzym besser (oder auch schlechter) funktioniert. Die vorgeschlagenen Veränderungen werden durch gezielte Mutationen in dem Gen vorgenommen, man geht zurück zu Schritt 7 und prüft, wie sich die Funktion verändert hat.
9. Ist man bei einem Enzym angekommen, das für eine technische Anwendung geeignet ist, beginnt der „Papierkram“: die Sicherheit muss überprüft werden, Zulassungen werden beantragt und ja, auch Patente werden eingereicht denn auch eine Universität, die schließlich aus Steuergeldern finanziert wird, will und muss die hohen Forschungskosten wieder hereinholen, wenn einmal ein marktfähiges Produkt herauskommt.
10. Aber selbst bei einem Patent ist noch nicht gesichert, dass ein Anwender im nächsten Jahr die Welt von allen PET-Flaschen befreit. Es ist ein langwieriger Prozess, der oft weitere Optimierungen und vor allem auch hohe Investitionen braucht. Die wichtigste Basis, die Grundlagenforschung, gerät dabei oft in Vergessenheit.
Struktur- und Funktionsanalyse
Mit der dreidimensionalen Struktur eines Proteins können Laien kaum etwas anfangen. Aber versuchen wir es mal:
die Struktur hat im unteren Teil Ähnlichkeiten mit anderen bakteriellen PETasen, die PET-Pulver hydrolysieren können. Ungewöhnlich sind die beiden Bänder (alpha-Helices) oben rechts, die als „Lid“ bezeichnet sind. Diesen „Deckel“ kennt man aus anderen Enzymen (aber nicht anderen PETasen!), bei denen er an der Bindung des Substrats beteiligt ist. Erste Experimente zeigen, dass sich der „Deckel“ positiv auf die Aktivität des neuen Enzyms auswirkt. Nimmt man ihn weg, wird PET wesentlich schlechter abgebaut.
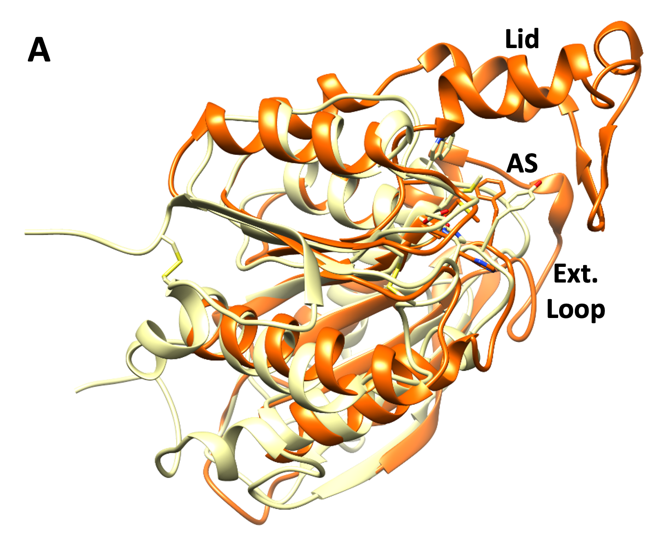
Struktur der PETase PET46 (Abbildung zur Verfügung gestellt von Wolfgang Streit, Universität Hamburg)
Wissenschaftler können oft aus Strukturen die Funktionsweise ableiten und durch Vergleiche manchmal sogar Vermutungen anstellen, wie ein Enzym noch effektiver arbeiten kann. Auch das kann man im Labor überprüfen: durch gezielte genetische Veränderungen wird ein modifiziertes Protein produziert und seine Leistung wird im Reagenzglas getestet.
Dass PET 46 am besten bei 70°C funktioniert war nicht überraschend – schließlich stammt der Organismus aus einer ziemlich heißen Umwelt.
… die unendlichen Weiten der Mikrobiologie …
„… es gibt wohl kaum ein Molekül, dass Mikroben nicht abbauen können.“ sagt Ruth Schmitz-Streit, die das neue Enzym zusammen mit Kollegen aus Hamburg und Düsseldorf gefunden hat. Man muss sie nur finden! Es gibt Millionen verschiedener Bakterien- und Archaeenarten – aber man kennt nur einen winziger Bruchteil davon durch Laborarbeiten. Was der Rest alles leisten könnte, weiss man nicht. Metagenome, Bioinformatik und gentechnische Methoden werden noch für viele Überraschungen sorgen – und zur Lösung vieler Probleme beitragen. Zur Anwendung ist es dann noch einmal ein großer Schritt, aber ohne die Grundlagenforschung geht gar nichts!
Auf der Seite der Universität Kiel gibt es eine Zusammenfassung der Arbeit.
Die Originalpublikation ist für Laien zwar kaum verständlich, sie ist aber hier frei zugänglich zu finden.
Anmerkung:
Ruth Schmitz-Streit ist auch Mitglied des Forschungskonsortiums SPP2141, über das BioWissKomm auf der Seite https://crispr-whisper.de/ berichtet. Schauen Sie auch da mal rein!
Eine weitere Methode, bei der sehr große Mengen an Sequenzdaten verarbeitet werden, finden Sie hier
Autor: Wolfgang Nellen, BioWissKomm
Abbildungen: Struktur PET 46, Wolfgang Streit, Universität Hamburg
Alle anderen Abbildungen: BioWissKomm, z.T. by Midjourney

