Analyse der Laborkurse bei der Roadshow in München
Experimente im Labor bezeichnet man auch als „Versuche“. Das bedeutet, dass man etwas versucht. Wir alle wissen, dass ein Versuch nicht immer erfolgreich ist. Ich kann versuchen, zehn Minuten die Luft anzuhalten. Ich kann auch versuchen einen Hefekuchen ohne Hefe zu backen. Dabei gehe ich von der Hypothese aus „zehn Minuten Luft anhalten kann ich, so viel Sauerstoff brauche ich nicht!“ und „Hefe ist nicht nötig, der Kuchen geht auch ohne auf und wird locker und lecker.“ Der Versuch zeigt dann, dass die Hypothese falsch war.
Bei Laborexperimenten ist das oft ähnlich. Eine Hypothese ist meist auf viel Vorarbeiten, Überlegungen und Diskussionen mit Kollegen begründet – und trotzdem erweist sie sich oft als falsch.
Ein Experiment kann übrigens eine Hypothese nur bestätigen, nie beweisen! Wenn man 1000 weiße Schwäne beobachtet hat, bestätigt das zunächst die Hypothese, dass alle Schwäne weiss sind. Wenn dann der 1001ste Schwan schwarz ist, dann ist die Hypothese falsifiziert!
Bei einem etablierten Experiment, das (fast) immer das erwartete Ergebnis liefert, sieht es etwas anders aus. Wenn es „nicht funktioniert“ hat man meistens etwas falsch gemacht oder es stimmte etwas nicht mit den Ausgangsmaterialien. Vorherige Experimente haben gezeigt, dass die Hypothese vermutlich richtig war. Jetzt geht es um die Fehlersuche!
Im schlimmsten Fall war bei allen vorherigen Experimenten was falsch – dann müsste insgesamt eine neue Hypothese aufgestellt werden.
Wir glauben, dass „fehlgeschlagene“ Experimente oft lehrreicher sind als solche, die so wie beschrieben, funktionieren. Dann muss man nämlich überlegen, warum es nicht wie erwartet geklappt hat! Solche Überlegungen, die Aufstellung von Hypothesen und Experimente zu ihrer Überprüfung, können durchaus in der Oberstufe, besonders in Leistungskursen, als Hausarbeiten gestellt oder auch in Klausuren eingesetzt werde. Sie fordern von Schülern kreatives Denken und gelernte Methoden in anderen Zusammenhängen einzusetzen (z.B. PCR, DNA-Sequenzierung, Gelelektrophorese usw.). Selbst wenn an einer Schule wenig Möglichkeiten zum Experimentieren gegeben sind, kann damit wissenschaftliches und vernetztes Denken gefördert werden.
Das CRISPR-Experiment
Unser CRISPR-Cas Experiment ist ausführlich hier beschrieben (auf Wunsch kann das PDF der Arbeit zugeschickt werden). Kurz gesagt: im Bakterium E. coli wird mit CRISPR-Cas das Gen für ß-Galaktosidase (lacZ) ausgeschaltet. Mit dem Enzym ß-Galaktosidase können Bakterien unter bestimmten Bedingungen blau angefärbt werden. Wird das Gen ausgeschaltet, bleiben die Bakterien weiss. Ebenso in einem Kontrollexperiment, in dem neben dem Cas9 Gen eine „falsche“ crRNA enthalten ist, mit der das lacZ Gen nicht erkannt wird. Das sieht dann üblicherweise so aus:
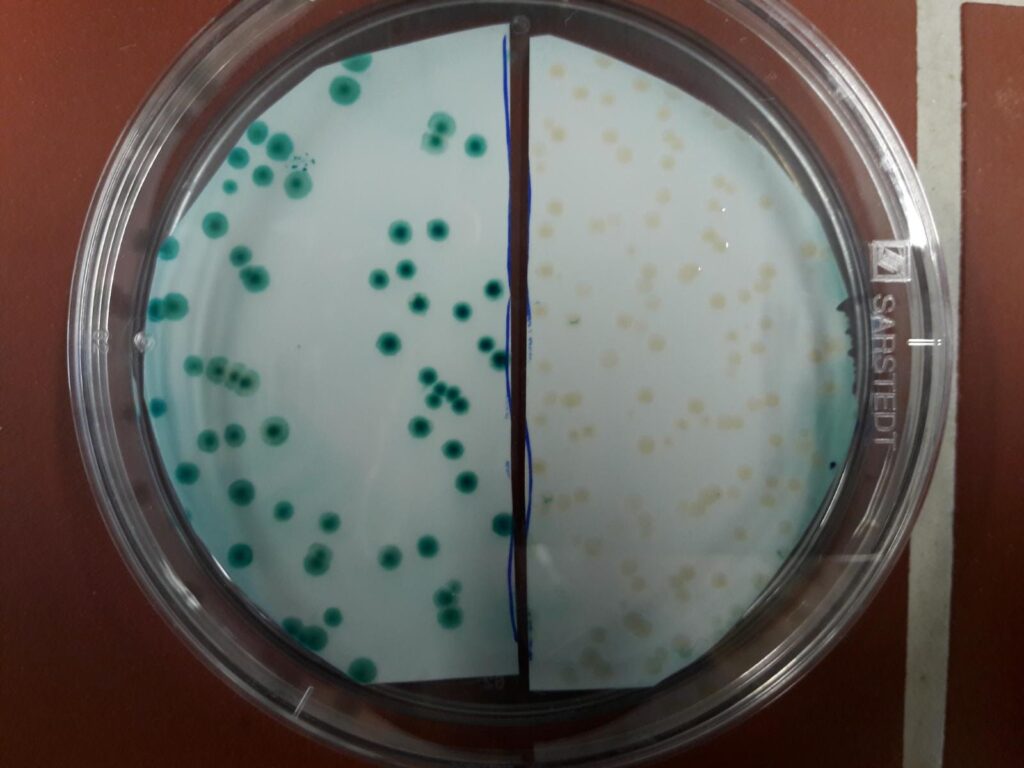
Wir haben dieses Experiment oft gemacht. Hier ist ein Beispiel von der Roadshow in Regensburg:
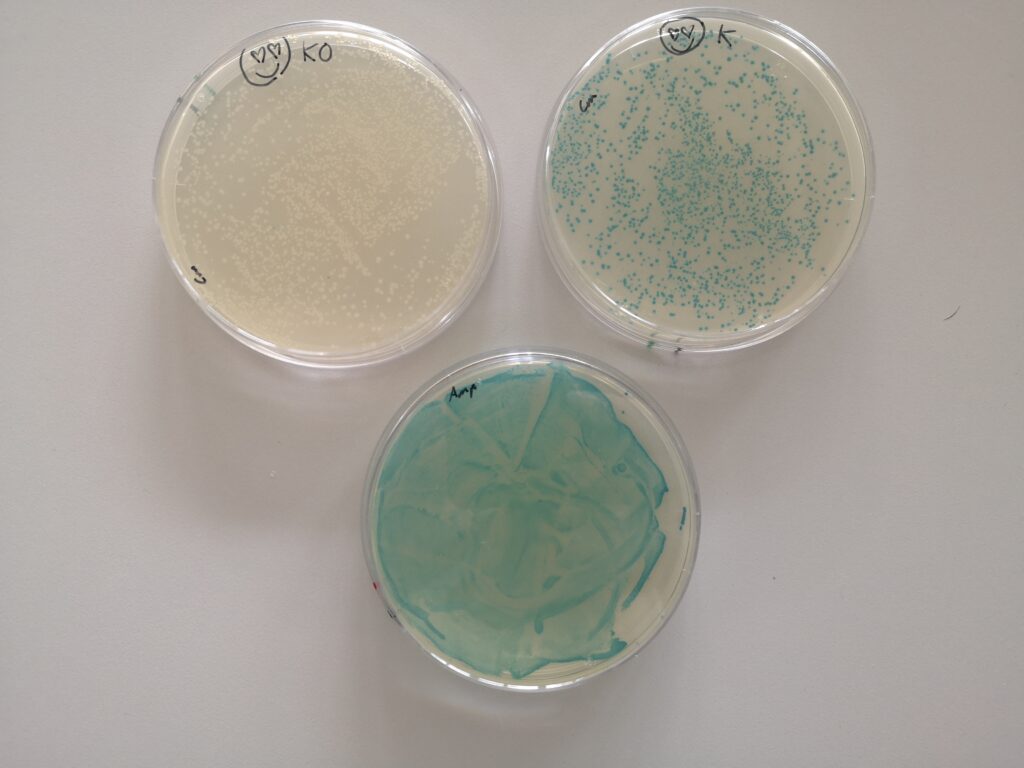
In München sah es dagegen so aus:
sowohl auf der Kontrollplatte als auch auf der Platte, auf der das lacZ Gen durch CRISPR-Cas zerschnitten sein sollte, waren alle Kolonien blau.
Das war bei allen Kursteilnehmern so. Wir konnten also nicht behaupten, dass alle Gruppen den gleichen Feler gemacht oder etwas verwechselt hatten!
Damit hatten wir zwar nicht das
gewünschte Ergebnis, aber jetzt wurde das Experiment erst richtig spannend! Denn nun muss man auf die Fehlersuche gehen!
Warum hat das nicht so funktioniert, wie es sollte?
Dazu kann man Hypothesen aufstellen, die überprüfbar sein müssen . Wird die Hypothese im Experiment nicht bestätigt, ist sie „falsifiziert“ und muss verworfen werden. Dann muss man sich eine neue Hypothese einfallen lassen.
Wie sieht es mit der Hypothese aus, dass ein böser Geist das Experiment verhext hat? Das kann man tatsächlich nicht wissenschaftlich ausschließen! Es gibt kein Experiment, mit dem man diese Vermutung falsifizieren kann. Deshalb wäre das keine wissenschaftliche Hypothese.
Aber hier sind ein paar echte, überprüfbare Hypothesen:
1. Ist der Schnitt im lacZ Gen erst später erfolgt?
Es dauert einige Zeit, bis die Bakterien das Cas9 Gen in RNA transkribiert und dann in Protein translatiert haben. Es könnte also sein, dass die Bakterien erst später weiss werden. Wenn man nun die blauen Zellen nach ein paar Tagen neu auf einer Agarplatte ausstreicht, sollten sie dann weiss sein.
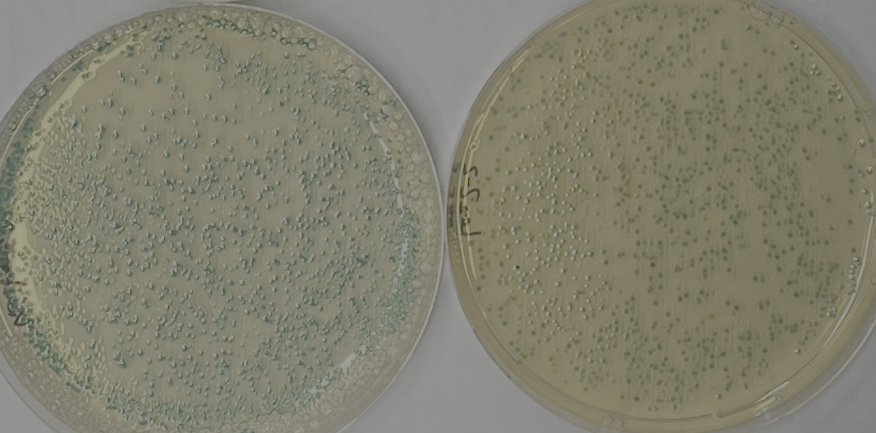
Andreas Brachmann von der LMU (Ludwig-Maximilians Universität München) hat mit den Teilnehmern ein paar Kolonien aus den Kursen ausgestrichen und das sah so aus:
Auch auf der frischen Platte waren alle Bakterien blau!
Schade, die Hypothese war damit falsifiziert und stimmte nicht!
2. Hatte das Cas9 Plasmid die crRNA verloren?
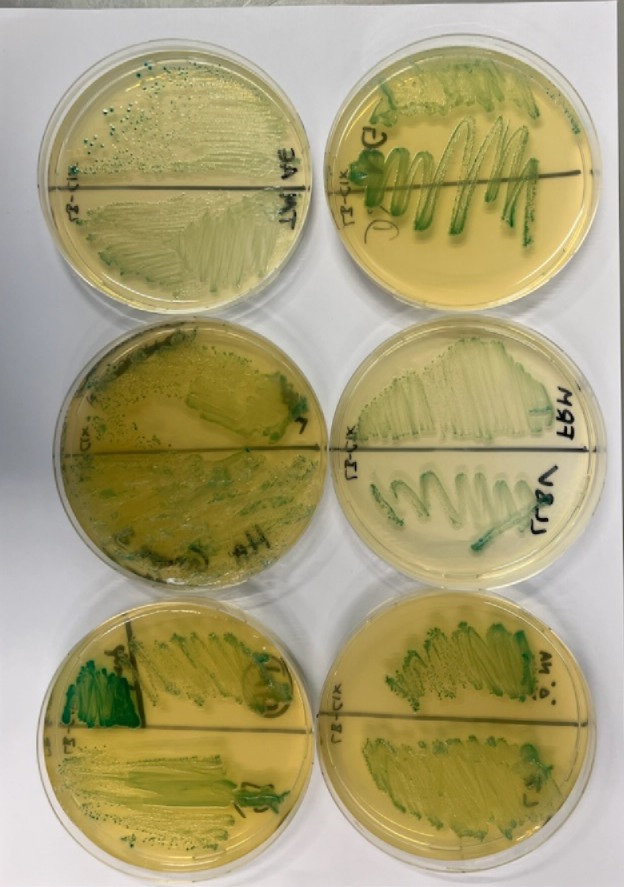
Wir hatten schon mal gesehen, das auf dem CRISPR-Plasmid die Sequenz für die Guide-RNA (der Steckbrief, um das lacZ-Gen zu finden und zu schneiden) verloren gegangen war (wie wir das gefunden haben, kann man hier nachlesen.). Vielleicht hatten wir zufällig ein solches Plasmid erwischt, als wir das für den Kurs vermehrt hatten? Auch das hat Andreas überprüft und die DNA mehrerer Plasmidpräparation sequenziert.
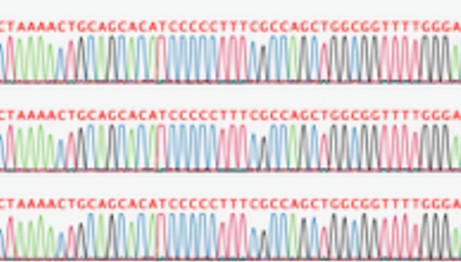
Man sieht bereits an den Farbmustern in Abb. 5a, dass alle gleich aussehen. Bei genauer Betrachtung findet man auch die vollständige Sequenz der crRNA. Alle sequenzierten Cas9 Plasmide hatten die erwartete Sequenz – nichts war verlorengegang. Auch diese Hypothese musste verworfen werden.
3. Hatten die Bakterien überhaupt das CRISPR-Cas Plasmid aufgenommen?
Das sollte schon so sein, denn schließlich waren sie auf dem Antibiotikum Chloramphenicol gewachsen und diese Resistenz konnte eigentlich nur von dem Cas9-Plasmid kommen! Aber wer weiss? Vielleicht war das Chloramphenicol nicht mehr gut und es gab keine Selektion auf die Resistenz?
Teilweise haben wir das mit dem ersten Kurs in München überprüft. In einem sehr einfachen Experiment hatten wir Bakterien von einzelnen Kolonien gepickt, die Zellen durch Hitze aufgebrochen und die freigesetzte DNA auf einem Agarosegel aufgetrennt. Dabei läuft die DNA des Bakterienchromosoms nur ein kurzes Stück ins Gel. Die Plasmide sind viel kleiner und wandern weiter. Dabei ist das Plasmid mit dem lacZ-Gen (ca. 6.675 bp) deutlich kleiner als das Plasmid mit dem Cas9 Gen und der crRNA (ca. 9.326bp). Dieses Experiment funktioniert nicht immer gut, weil in den kleinen Bakterienkolonien nicht immer genug DNA drin ist. Abb. 6 zeigt das Ergebnis und aus welchen Klonen die DNA stammt.
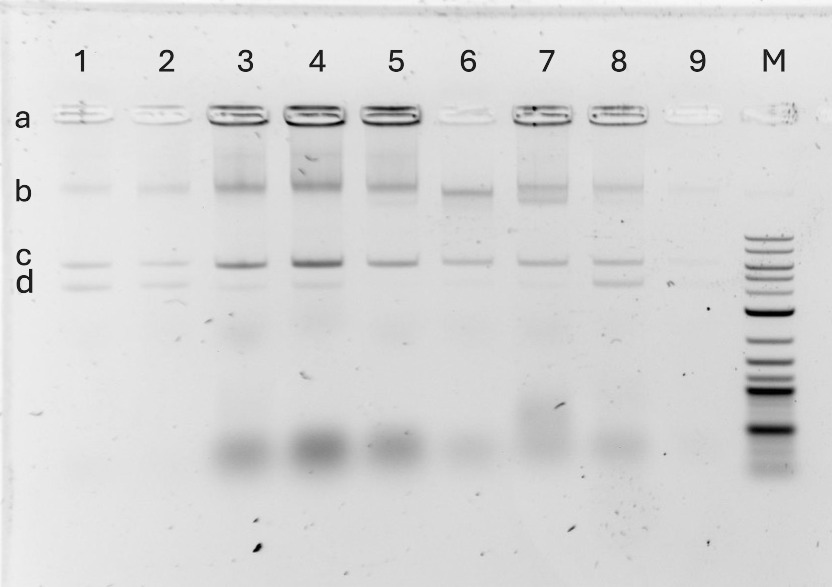
Die Proben wurden oben in der Tasche (a) aufgetragen und wanderten im elektrischen Feld nach unten zum Plus-Pol, weil DNA negativ geladen ist.
In der Position b läuft die chromosomale DNA der Bakterien. Die ist riesig groß und man kann die Größe nicht richtig bestimmen. In der Spur 6 ist sie etwas kleiner und in Spur 7 gibt es zwei Banden – das können wir im Moment auch nicht richtig erklären (ein weiteres Rätsel!).
In der Position c läuft das große Plasmit mit dem Cas9-Gen und der crRNA (9326 Basenpaare), in der Position d läuft das kleinere Plasmid mit dem lacZ-Gen (6675 Basenpaare).
In den Spuren sind aufgetragen:
1 Kontrolle („falsche“ crRNA)
2 Kontrolle („falsche“ crRNA)
3 lacZ („richtige“ crRNA)
4 lacZ („richtige“ crRNA)
5 lacZ („richtige“ crRNA), ein besonders stark blauer Klon
6 lacZ („richtige“ crRNA)
7 lacZ („richtige“ crRNA)
8 Kontrolle („falsche“ crRNA)
9 Kontrolle („falsche“ crRNA)
10 Größenmarker (nur zur „Dekoration“, der sagt uns hier nicht viel!
In Spur 3 und 4 sieht man sehr schwach auch noch das lacZ Plasmid. Das sollte nicht so sein denn mit der richtigen crRNA sollte es geschnitten und abgebaut werden.
Mit etwas Phantasie sieht man es auch in den Spuren 6 und 7.
Ob es in Spur 5 vorhanden ist, kann man schwer sagen. Da müsste man genauer nachsehen! Aber gerade dieser Klon war sehr stark blau und sollte das Plasmid enthalten!
In Spur 8 sieht man wieder, wie in der Kontrolle zu erwarten, beide Plasmide.
In Spur 9 sieht man nichts. Da ist einfach zu wenig DNA isoliert worden!
Auch die Hypothese, dass das Cas9 Plasmid nicht aufgenommen wurde, kann also verworfen werden.
Zusammenfassung
Zusammenfassend können wir feststellen, dass sowohl das Cas9 Plasmid mit der „richtigen“ crRNA als auch das mit der „falschen“ crRNA aufgenommen wurde (die beiden Plasmide sind in ihrer Größe nicht unterscheidbar).
Die crRNA war in dem großen Plasmid korrekt vorhanden.
Auch nach längerer Inkubation wurden die Klone nicht weiß.
Alle unsere Hypothesen konnten falsifiziert werden und waren folglich nicht richtig
Wir sind trotzdem klüger als vorher, denn wir wissen nun, was nicht der Grund dafür war, dass das Ergebnis anders als erwartet ausfiel!
Was nun?
Es gibt aber bestimmt noch weitere Hypothesen um das Rätsel zu lösen, wir hatten aber keine Zeit, über alle Möglichkeiten nachzudenken. Vielleicht fällt euch ja noch etwas ein – und ein Vorschlag, wie man eure Hypothese überprüfen kann? Schreibt uns direkt auf dieser Seite in den Kommentaren oder an info@biowisskomm.de!
Wenn es ein paar Vorschläge gibt, wollen wir versuchen, noch etwas Geld aufzutreiben (solche Experimente kosten Zeit und Geld!) und wir werden versuchen Andreas Brachmann zu überreden, die Experimente in München zu machen!
Wir freuen uns auf eure Vorschläge!
