Wie man Gene zum Schweigen bringt
Grundlagen und Anwendung der RNA-Interferenz in Medizin und Landwirtschaft
Update am 6.7.2024 im Abschnitt „Anwendungen in der Landwirtschaft“
RNA Interferenz (RNAi) ist, wenn man so will, ein Teilgebiet der Epigenetik. Kleine RNAs (siRNA) wirken auf mRNAs. Sie verursachen (meist) ihren Abbau und regulieren so die Genexpression. Gentechnisch wird RNAi in der Grundlagenforschung als „Gene silencing“ oder „Knock down“ angewendet: dabei wird die genetische Information eines Gens nicht verändert, seine Expression aber zu einem großen Teil (nicht vollständig!) abgeschaltet. Es gibt inzwischen mehrere zugelassene Medikamente auf RNAi-Basis und erfolgversprechende Ansätze zur Schädlingsbekämpfung in der Landwirtschaft.
Um das Jahr 1990 wurde ein seltsames Phänomen beobachtet: man versuchte in Petunien die Blütenfarbe intensiver zu machen, indem man gentechnisch die Farbstoffproduktion verstärkte. Statt dessen fiel der Farbstoff teilweise vollständig aus.

Wie das funktionierte wusste man nicht, erfand aber den Namen „Co-suppression“. Man vermutete, dass die Überexpression des Farbgens zu „aberranter RNA“ führte – hatte aber keine Ahnung, was „aberrant“ sein könnte. Andere machten ähnliche Beobachtungen und nannten es „Post transcriptional gene silencing“ oder PTGS. Und dann gab es noch die Pilzforscher, die es „Quelling“ nannten.
Über fast 10 Jahre wurde diskutiert, verglichen und experimentiert um vielleicht eine gemeinsame Erklärung zu finden.
Antisense RNA
Wenn ein Gen aktiv wird, wird in der Regel nur ein Strang der doppelsträngigen DNA zu mRNA abgelesen (transkribiert). Der andere Strang würde „keinen Sinn“ machen (anti-sense) und könnte nicht in ein Protein übersetzt werden. Außerdem müsste am anderen Ende des Gens ein Promotor sitzen, der die Transkription anstößt.
Wenn es beide, eine „sense“ und eine „antisense“ RNA gäbe, dann könnten sie, ähnlich wie die DNA, ein doppelsträngiges Hybrid bilden. Das wäre ein weiteres Problem, weil doppelsträngige RNA nicht in Protein übersetzt werden kann.
Aufgrund dieser Überlegung und einiger seltenen und seltsamen Beispiele aus der Natur, kamen Wissenschaftler auf die Idee, Gene über antisense RNA auszuschalten. (Später stellte sich heraus, dass dieser Mechanismus wohl auch bei den Petunien eine Rolle spielte.)
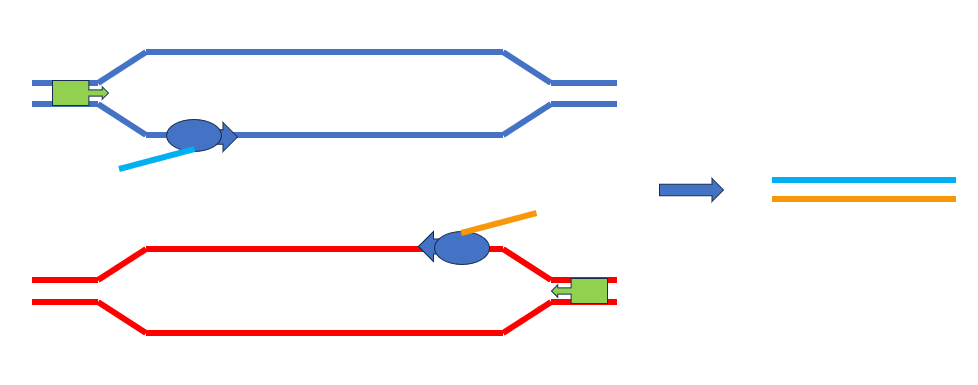
In einer künstlichen Konstruktion wird die gleiche DNA-Sequenz mit einem Promotor auf der rechten Seite versehen. Dieser dirigiert die RNA-Polymerase auf dem oberen Strang nach links. Es wird die orange RNA synthetisiert.
Die hellblaue und die orange RNA sind komplementär, d.h. sie können, wie die DNA, einen Doppelstrang bilden. Die hellblaue RNA kann nicht in Protein übersetzt werden, das Gen ist „stillgelegt“.
Erst später fand man heraus, dass der RNA Doppelstrang in kleine Teile von etwa 25 Basenpaaren zerschnitten wird.
(Grafik: © BioWissKomm)
Sie konstruierten künstliche „Gene“, bei denen den Promotor am „falschen“ Ende saß und brachten diese zusätzlich zum vorhandenen richtigen Gen in ihren Versuchsorganismus ein. In vielen Fällen funktionierte das tatsächlich: das Gen war ganz, oder zumindest teilweise ausgeschaltet!
Unerwartet war jedoch, dass man nicht etwa die doppelsträngige RNA fand, sondern gar nichts! Sowohl die sense als auch die antisense RNA waren verschwunden. Man spekulierte zwar, warum das so sein könnte, wichtiger war jedoch, dass es funktionierte und man so ganz gezielt in die Expression von Genen eingreifen konnte. Es gab einen „Antisense-Hype“ – aber ohne zu wissen, was wirklich passiert, war das nict so gut.
Wo ist die RNA geblieben?
Als Andrew Fire und Craig Mello 1998 eine Arbeit zur „RNA-Interferenz“ publizierten war die Zeit reif und innerhalb von wenigen Jahren war nicht nur ein großer Teil des Rätsels gelöst, sondern der ganze Mechanismus im Wettlauf vieler Labore aufgeklärt. Auch die Frage, wo denn nun die RNA abgeblieben war, wurde 1999 endlich geklärt. Man fand kleine RNAs von nur 25 Nukleotiden Länge, die offensichtlich Bruchstücke der mRNA (sense RNA) und der antisense RNA waren. Man nannte sie siRNA (small interfering RNA). Bis dahin hatte man sich mit solchem „Kleinkram“ kaum abgegeben und ihn für „Dreck“ gehalten, der bei der Präparation entstanden war.
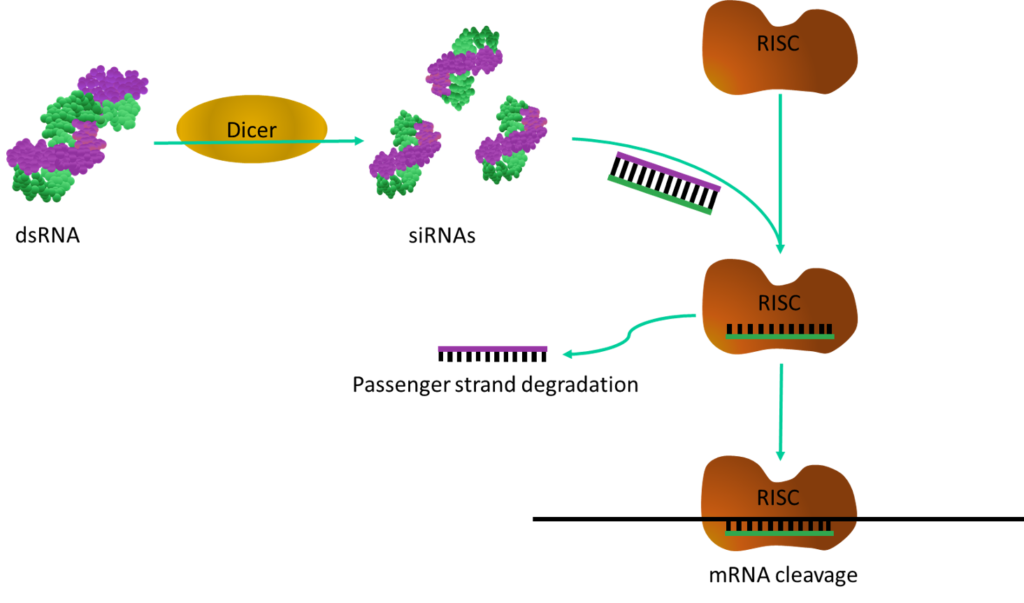
© Fraunhofer IME Maurice Pierry (mit freundlicher Genehmigung)
Doppelsträngige RNA wird vom Enzym Dicer erkannt und in ca. 25 Nukleotide lange doppelsträngige Bruchstücke zerlegt. Die werden dann an einen Proteinkomplex weitergereicht, dass als RISC bezeichnet wird. Die kurzen Doppelstränge werden getrennt. Einer davon verbleibt im RISC. Der Komplex erkennt, mit Hilfe der kleinen RNA, die entsprechende mRNA und zerschneidet sie. Das entsprechende Gen ist damit inaktiviert, ohne dass das Gen selbst verändert oder zerstört wird. Weil die Inaktivierung meist nicht zu 100% erfolgt, bezeichnet man das als „Knock-down“ (wird ein Gen völlig zerstört ist das ein „Knock-out“.)
Die siRNAs können aber noch mehr! Wenn sie an eine mRNA gebunden haben, muss diese nicht immer zerschnitten werden. Die siRNA kann von dem RdRP-Enzym (RNA dependent RNA polymerase) entlang der mRNA verlängert werden. Es entsteht wieder ein längerer RNA Doppelstrang, der von Dicer in weitere siRNAs zerschnitten wird. Dabei wird die mRNA auch zerstört, zusätzlich werden aber noch viel mehr sekundäre siRNAs produziert, die den mRNA-Abbau verstärken.
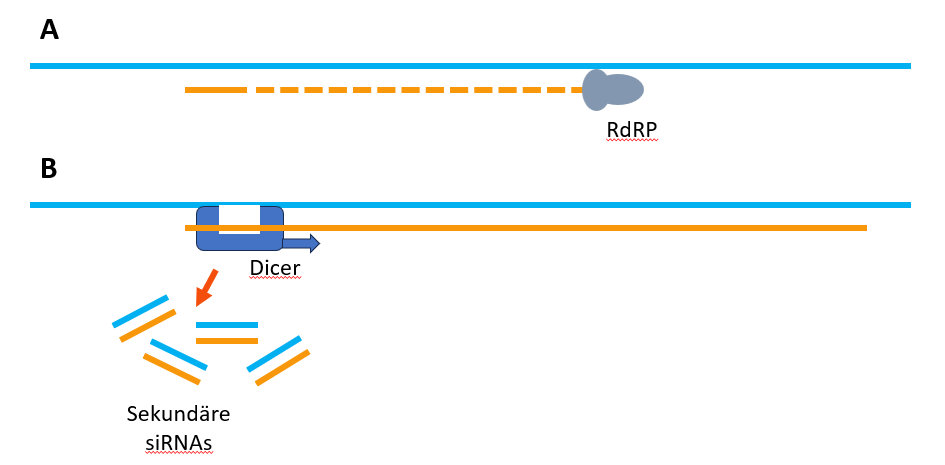
B die entstandene doppelsträngige RNA wird von Dicer in viele sekundäre siRNAs zerlegt. Diese können wiederum als Primer dienen oder an RISC binden und mRNAs einfach zerschneiden. (Grafik: © BioWissKomm)
Wofür ist das gut?
Ähnlich wie das CRISPR-Cas System in Prokaryoten kann die RNA Interferenz zur Virenabwehr dienen. Auch einige Gene werden damit reguliert. Besonders eindrucksvoll (vor allem weil meine Arbeitsgruppe selbst daran gearbeitet hat ????, ist die Kontrolle von Retrotransposons.
Retrotransposons sind ehemalige Viren, die sich im Genom fast aller Eukaryoten eingenistet haben. Wie alle „guten“ Parasiten, haben sie einen Deal mit ihrem Wirt verhandelt: sie schädigen ihn nicht zu stark und der Wirt toleriert sie dafür. Ein großer Schaden, den Retrotransposons anrichten können: sie vermehren sich und können sich selbst in verschiedene Stellen des Wirtsgenoms einbauen (sogenannte „springende Gene“). Das ist natürlich gefährlich denn so können sie wichtige Gene des Wirts unterbrechen und funktionslos machen. Zum „Deal“ zwischen Wirt und Parasit gehört die „Vereinbarung“, dass das Retrotransposon unbehelligt im Genom sitzen, aber nicht springen darf. Wie verhindert die RNA Interferenz das Springen?
Retrotransposons tragen meistens an beiden Enden ihres Genoms einen Promotor denn beide RNA Stränge werden gebraucht. Es entsteht also eine doppelsträngige RNA – und die wird von Dicer erkannt und zerschnitten.
Wie kann man das nachweisen?
In unserem Modellorganismus kann man gentechnisch das Gen für Dicer zerstören. Und was passiert dann?
Die Kontrolle über das Retrotransposon geht verloren und es fängt wieder an, im Genom zu springen und sich zu vermehren! Das ist für den Organismus nicht gut, weil es zu Mutationen führt.
Ist das im „richtigen Leben“ nützlich?
Wie oben gesagt kann eine kleine siRNA in einer Zelle vermehrt werden, wenn sie eine Ziel-RNA findet. Man braucht also nicht viel, um ein „Gen-Knock-down“ zu erzielen: kleine Mengen an synthetischen siRNAs reichen völlig aus.
Die erste Anwendung gab es mit Patisiran, einer synthetischen siRNA, die gegen Transthyretin-Amyloidose eingesetzt wird. Dabei handelt es sich um eine (oft) erbliche Erkrankung, die durch ein mutiertes Transthyretin-Gen verursacht wird und zu Schädigung des Herzmuskels und des Nervensystems führt. Es folgten weitere siRNA Medikamente gegen Porphyrie (Fehler im Eisenstoffwechsel), hohe Cholesterinproduktion und andere. In der Medizin hat diese Grundlagenforschung also bereits zu beachtlichen Erfolgen geführt!
In der Pipeline: Anwendungen in der Landwirtschaft
Weitere Anwendungen werden intensiv erforscht. Eine interessante Idee ist der Einsatz von siRNAs als Spray für den Pflanzenschutz.
Weil man die üblichen Schadinsekten in der Landwirtschaft kennt, kann man lebenswichtige Zielgene auswählen und ein Knock-down mit RNA Interferenz versuchen. Im Prinzip funktioniert das bereits und man hat ein Verpackungsmaterial entwickelt, das die siRNA schützt und die Aufnahme durch die Insekten erleichtert. Nun müssen Feldversuche durchgeführt werden was in Deutschland leider nahezu unmöglich ist. Voraussichtlich wird man das im Ausland machen müssen.
Ein großer Vorteil dieser Methode: im Gegensatz zu chemischen Pestiziden können siRNAs sehr spezifisch gegen einen Schadorganismus gerichtet werden. „Kollateralschäden“ bei Nutzinsekten sind praktisch ausgeschlossen, weil sich die Zielgene unterscheiden. Beim „falschen“ Organismus findet die siRNA kein Ziel und es passiert nichts. Zudem wird RNA schnell abgebaut (sie muss gut verpackt werden, damit sie nicht zu schnell abgebaut wird!). Ein langer Verbleib in der Umwelt oder andere, langfristige ökologische Schäden sind deshalb nicht zu erwarten. Um das zuverlässig zu untersuchen, sind aber kontrollierte Experimente im Freiland erforderlich.
Wenn erfolgreich, könnte RNAi den Pflanzenschutz in der Landwirtschaft wesentlich umweltfreundlicher machen. Wenn man es genau betrachtet, handelt es sich um eine biologische Methode bei der nicht gentechnisch ins Genom eingegriffen wird. Sie sollte also auch im Bio-Landbau erlaubt sein, der bisher auf viele chemische Insektizide verzichten und nicht selten mit entsprechenden Ernteausfällen rechnen muss.
Update
Ein RNAi-Spray gegen den Kartoffelkäfer ist in den USA inzwischen zugelassen und wird (hoffentlich) den Bauern helfen, dieses Schadinsekt zu bekämpfen.
Gleichzeitig erkennt man aber bereits Wege, wie die Insekten Resistenzen gegen RNAi entwickeln. Dabei wird nicht etwa das Zielgen mutiert, sondern gleich der ganze RNAi-Mechanismus lahmgelegt.
Überraschend ist das nicht: Evolution ist erfinderisch und manchmal sehr schnell. Bei allen Pestiziden werden (ebenso wie wir es von Antibiotika kennen) füher oder später Resistenzen auftreten. So funktioniert Evolution nun einmal. Die Herausforderung ist, die Resistenzbildung so lange wie möglich zu verzögern. Dazu hilft ein kluges Pestizid-Management und sparsamer Gebrauch!

Autor: W. Nellen, BioWissKomm
Titelbild: created by Midjourney © BioWissKomm

